Boletins de notícias LNCC

O LNCC Notícias é um boletim digital online, de acesso público e periodicidade mensal, para divulgar as atividades e notícias do Laboratório Nacional de Computação Científica (LNCC/MCTIC). A reprodução parcial ou total das notas é autorizada, desde que acompanhada das devidas referências e créditos da publicação, indicando o link para a página.
O Laboratório Nacional de Computação Científica - LNCC (unidade de pesquisa vinculada ao Ministério da Ciência, Tecnologia e Inovações - MCTI) anuncia a programação de abril do ciclo de palestras da pós-graduação em Modelagem Computacional. Assim como nas edições anteriores, as palestras são gratuitas e abertas ao público em geral através de webinars do aplicativo Zoom, sendo também transmitidas ao vivo pelo canal do LNCC no YouTube.
Confira a programação para o mês de abril:
05/04 às 14h
DockThor-VS: Uma plataforma computacional para triagem virtual em larga escala visando alvos moleculares de SARS-COV-2 e suas variantes genômicas, com Laurent E. Dardenne, pesquisador do Laboratório Nacional de Computação Científica (LNCC/MCTI)
Variações não sinônimas de SARS-CoV-2 e impacto no desenvolvimento de fármacos e vacinas, com Marisa F. Nicolás, pesquisadora do Laboratório Nacional de Computação Científica (LNCC/MCTI)
Link para inscrição: https://us02web.zoom.us/webinar/register/WN_dFURhYeVRhKd3GUAaKRzkA
12/04 às 14h
Série Alumni - Modelagem do Escoamento de Espumas em Meios Porosos, com Thiago Quinelato, pesquisador de Pós-doutorado na Universidade Federal de Juiz de Fora (UFJF) e ex-aluno do LNCC.
Link para inscrição: https://us02web.zoom.us/webinar/register/WN_QIl-nkCPSzu9EoJQ8qz4Yg
19/04 às 14h
Breve História da Supercomputação, com Stephan Stephany, pesquisador titular do Instituto Nacional de Pesquisas Espaciais (INPE/MCTI).
Link para inscrição: https://us02web.zoom.us/webinar/register/WN_2QgCUI63Tze2Td5mnzSoqw
26/04 às 14h
Colóquio LNCC - Desigualdades Matriciais Lineares Diferenciais, com José C. Geromel, professor titular da Faculdade de Engenharia Elétrica e de Computação da UNICAMP.
Link para inscrição: https://us02web.zoom.us/webinar/register/WN_1Z4KolNkSH2_jP3yG9U3_Q

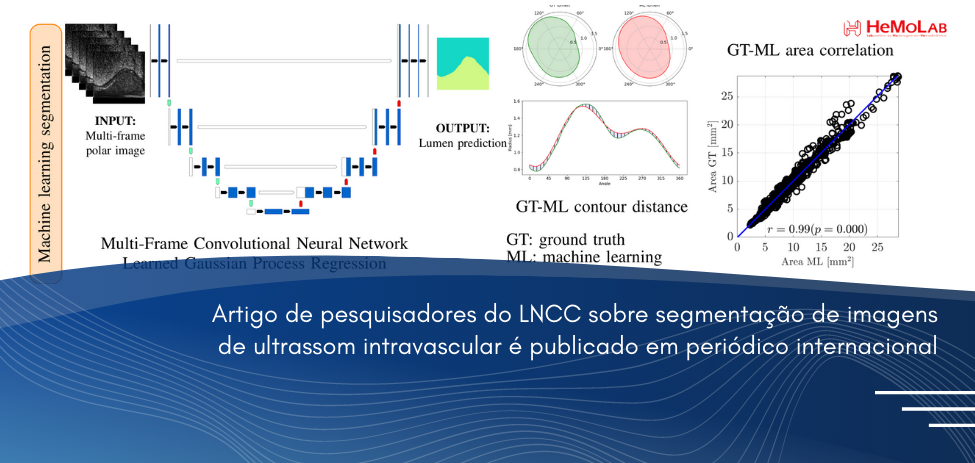
O trabalho de pesquisa intitulado Automated lumen segmentation using multi-frame convolutional neural networks in intravascular ultrasound datasets foi recentemente publicado no periódico internacional European Heart Journal - Digital Health. A plataforma, da Sociedade Européia de Cardiologia tem acesso aberto e foco específico em saúde digital na medicina cardiovascular. O artigo científico apresenta uma metodologia computacional completamente automática que permite realizar a segmentação de estruturas anatômicas de interesse, em particular o contorno do lumen arterial, em imagens de ultrassom intravascular (IVUS). A metodologia se baseia na combinação de um algoritmo para registro das imagens, uma rede neural convolucional adaptada para o tipo de dado utilizado, e um algoritmo de regressão não paramétrico baseado no conceito de processos Gaussianos. A acurácia apresentada pelo método nos permite concluir que esta classe de metodologia permitirá transferir ferramentas da computação científica para a rotina clínica, reduzindo o custo envolvido na avaliação manual desta classe de imagens.
O trabalho foi coordenado pelo professor e pesquisador do LNCC, Pablo Javier Blanco em colaboração com os membros do Laboratório de Modelagem em Hemodinâmica HeMoLab/LNCC. Também colaboraram com a pesquisa o Hospital Albert Einstein de São Paulo e o Medstar Washington Hospital Center (Virginia, USA).
O artigo publicado está disponível em https://academic.oup.com/ehjdh/article/1/1/75/5998645?login=true.
As atividades centrais do HeMoLab (Hemodynamics Modeling Laboratory) envolvem modelagem e simulação numérica de sistemas fisiológicos, mais especificamente o sistema cardiovascular. O desenvolvimento de softwares é uma preocupação constante do grupo, visando a popularização de ferramentas de modelagem e simulação e a facilitar seu emprego em problemas reais de larga escala. Conheça mais em http://hemolab.lncc.br/
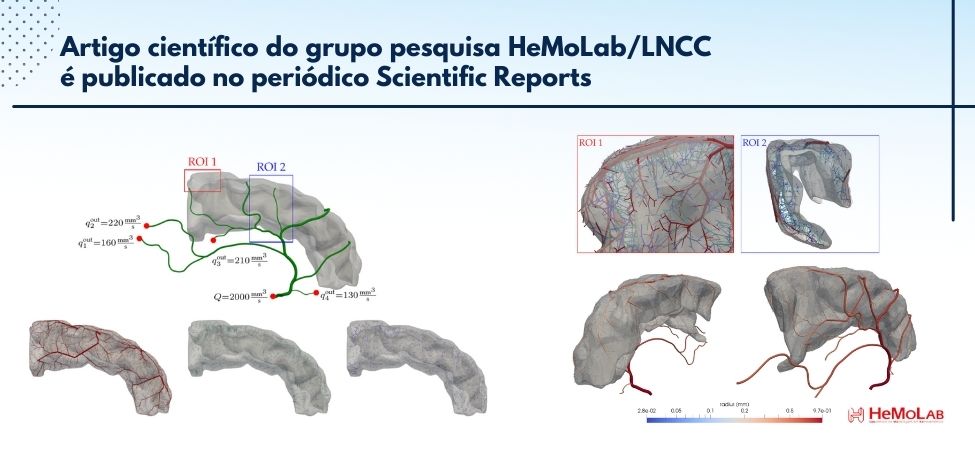
Um reconhecido periódico científico da área interdisciplinar, a revista Scientific Reports, publicou recentemente uma pesquisa conduzida por cientistas do Laboratório Nacional de Computação Científica - LNCC/MCTI em colaboração com o Auckland Bioengineering Institute (University of Auckland, Nova Zelândia). A pesquisa é mais uma iniciativa do Laboratório de Modelagem em Hemodinâmica - HeMoLab/LNCC, coordenado pelo professor e pesquisador Pablo Javier Blanco.
O trabalho de pesquisa intitulado "Adaptive constrained constructive optimisation for complex vascularisation processes" propõe um conjunto de ferramentas computacionais para modelar o processo de construção de redes vasculares em cenários complexos, tanto do ponto de vista das restrições angiogênicas envolvidas como da geometria anatômica onde se constroem tais redes vasculares. Este desenvolvimento permitirá construir modelos visando conectar a circulação sanguínea nas escalas espaciais observáveis nas imagens médicas com as escalas microcirculatórias e a fisiologia característica de cada órgão.
O artigo pode ser acessado em https://www.nature.com/articles/s41598-021-85434-9
As atividades centrais do HeMoLab (Hemodynamics Modeling Laboratory) envolvem modelagem e simulação numérica de sistemas fisiológicos, mais especificamente o sistema cardiovascular. O desenvolvimento de softwares é uma preocupação constante do grupo, visando a popularização de ferramentas de modelagem e simulação e a facilitar seu emprego em problemas reais de larga escala. Conheça mais em http://hemolab.lncc.br/
O Laboratório Nacional de Computação Científica - LNCC lamenta profundamente o falecimento do professor Artur Ziviani, ocorrida no último dia 24 de março de 2021 de decorrência de complicações da COVID-19.
Artur Ziviani graduou-se em Engenharia Eletrônica pela Escola Politécnica da UFRJ em 1998. Obteve o título de Mestre em Engenharia Elétrica (com ênfase em Teleinformática) pela COPPE/UFRJ em 1999 e o título de Doutor em Systèmes Informatiques pela Université Paris VI, Sorbonne Universités, França, em dezembro de 2003. Durante o ano letivo 2003-2004 lecionou na Université Paris VI. Foi pesquisador visitante do INRIA, França, por 5 meses entre 2008 e 2009. Professor Ziviani era membro afiliado da Academia Brasileira de Ciências desde 2013, membro Senior IEEE desde 2011 e membro Senior da Association for Computing Machinery (ACM). Professor Artur foi contemplado duas vezes no programa Jovem Cientista do Nosso Estado da FAPERJ. Além do longo currículo como pesquisador, Prof. Ziviani teve fundamental importância na nova colaboração entre LNCC e Inria (França). Nos últimos 22 meses professor Artur esteve à frente da Coordenação de Pós-Graduação do LNCC.
Em sua renomada carreira, destacou-se profissionalmente com várias premiações:
2016 - Menção honrosa (entre os 4 melhores artigos completos) do SBBD'2016 por artigo em co-autoria com Daniel N. R. da Silva, Klaus Wehmuth, Carla Osthoff e Ana Paula Appel, SBC.
2017 - Melhor artigo do WPerformance 2017 - Recomendação de Conteúdo e Desempenho de Sistemas de Cache - em co-autoria com R. G. M. de Freitas, D. S. Menasché e C. Delgado., SBC.
2018 - Menção honrosa por artigo no WGRS 2018, SBRC 2018, em co-autoria com A. T. Oliveira, B. JC. A. Martins, A. T. A. Gomes, M. F. Moreno e A. B. Vieira, SBC.
2018 - Menção honrosa (entre os 3 melhores artigos completos) do WPerformance 2018 por artigo em co-autoria com G. Carnivali, A. B. Vieira, P. Esquef, SBC.
2018 - Melhor artigo do WBlockchain 2018, SBRC 2018, - Análise da Rede de Transações do Ethereum - em co-autoria com J. G. Z. Mascarenhas e A. B. Vieira., SBC.
Ziviani deixa esposa e dois filhos, além de um vazio em todos que com ele tiveram a oportunidade de conviver. Uma perda irreparável para o LNCC, uma perda irreparável para todos os seus amigos que tiveram a oportunidade de vivenciar sua generosidade.
A comunidade do LNCC lamenta profundamente a perda e deseja forças a família para superar esse momento de profunda tristeza.

O artigo do Grupo de Modelagem Molecular em Sistemas Biológicos foi publicado pela Scientific Reports.
Durante a pandemia do novo Coronavírus, o Laboratório Nacional de Computação Científica - LNCC (unidade de pesquisa vinculada ao Ministério da Ciência, Tecnologia e Inovações - MCTI) tem desempenhado um importante papel para a comunidade científica mundial, ao desenvolver pesquisas e projetos que utilizam a computação de alto desempenho e a capacidade do supercomputador Santos Dumont para achar respostas mais eficientes na luta contra a Covid-19. E uma dessas respostas, vem do Grupo de Modelagem Molecular em Sistemas Biológicos - GMMSB, que teve o artigo Drug Design and Repurposing with DockThor-VS Web Server focusing on SARS-CoV-2 Therapeutic Targets and their Non-Synonym Variants publicado pelo periódico científico internacional Scientific Reports, que pertence à editora Nature Research.
A pesquisa, coordenada pelo prof. Laurent Dardenne, biofísico e líder do de pesquisa GMMSB do LNCC, apresenta os desenvolvimentos implementados no servidor web DockThor-VS para fornecer uma plataforma de triagem virtual (Virtual Screening, VS) com estruturas em três dimensões de potenciais alvos terapêuticos do SARS-CoV-2 incorporando informações genéticas sobre mutações relevantes.
Na plataforma é possível fazer estudo de reposicionamento de fármacos com vários alvos e variantes do vírus, como a variante brasileira, sul-africana e inglesa. Toda a comunidade científica mundial pode fazer pesquisa com essa plataforma, que está vinculada ao supercomputador Santos Dumont, ressalta Laurent Dardenne.
O portal vem sendo utilizado por pesquisadores do mundo inteiro, com média diária de dezenas de submissões. Já são mais de 3.477 trabalhos esse ano. No ano passado inteiro foram 15.635 submissões. O servidor web facilita a realização de experimentos de triagem virtual objetivando estudos de reposicionamento de fármacos através da disponibilização de uma biblioteca contendo os medicamentos, aprovados pelo Food and Drug Administration (FDA), disponíveis no mercado. Atualmente, o DockThor-VS fornece estruturas 3D prontas para triagem virtual para seis alvos terapêuticos de SARS-CoV-2: Nsp3 (PLpro), Nsp5 (Mpro, 3CLpro), Nsp12 (RdRp), Nsp15 (NendoU), proteína N e proteína Spike. São disponibilizadas no portal as estruturas 3D para a sequência genômica selvagem e para as variantes genômicas não sinônimas mais relevantes (fruto de um estudo envolvendo mais de 40 mil genomas de SARS-CoV-2). No total estão disponibilizadas 45 estruturas 3D até o momento, ressalta Isabella Guedes, primeira autora do artigo e pesquisadora de pós-doutorado do LNCC. O estudo das variantes genômicas envolveu uma colaboração com a profa. Marisa Nicolás, geneticista especializada em bioinformática, também pesquisadora do LNCC e associada ao Laboratório de Bioinformática (Labinfo/LNCC).
Recentemente, o grupo de pesquisa também publicou outro artigo no mesmo periódico científico com a descrição da ferramenta de inteligência artificial que prevê a potência das moléculas nos estudos de triagem virtual. As publicações embasam cientificamente o uso do portal e as metodologias de triagem virtual. Nesse momento, quando todo mundo está preocupado com as variantes do vírus, é importante saber se as moléculas pesquisadas terão efeito também sob estas variantes, como as de Manaus e da Inglaterra, ou se haverá algum mecanismo de resistência, por exemplo, pontua Dardenne.
Além do professor Dardenne, da professora Marisa Nicolás e da pós-doc Isabella Guedes, o trabalho contou ainda com a colaboração da pós-doc Karina B. dos Santos, dos alunos da pós-graduação Leon S. C. Costa e Ana L. M. Karl, dos pesquisadores do LNCC Fábio L. Custódio e Helio J. C. Barbosa, e os tecnologistas do SINAPAD Iury M. Teixeira, Marcelo M. Galheigo e Vivian Medeiros. Também participaram da pesquisa ex-alunos do LNCC, Eduardo Krempser e Gregório Kappaun, atualmente pesquisadores da Fundação Oswaldo Cruz e do Instituto Federal Fluminense - IFF (Campus Macaé), respectivamente.
O trabalho foi realizado com financiamento das agências de fomento à pesquisa FAPERJ, CAPES e CNPq. O DockThor-VS está disponível gratuitamente em www.dockthor.lncc.br e os artigos podem ser acessados nos links abaixo:
Guedes, Isabella A., Leon S. C. Costa, Karina B. dos Santos, Ana L. M. Karl, Gregório K. Rocha, Iury M. Teixeira, Marcelo M. Galheigo, Vivian Medeiros, Eduardo Krempser, Fábio L. Custódio, Helio J. C. Barbosa, Marisa F. Nicolás, and Laurent E. Dardenne. 2021. Drug Design and Repurposing with DockThor-VS Web Server Focusing on SARS-CoV-2 Therapeutic Targets and Their Non-Synonym Variants. Scientific Reports 11(1):5543. doi: 10.1038/s41598-021-84700-0.
Guedes, Isabella A., André M. S. Barreto, Diogo Marinho, Eduardo Krempser, Mélaine A. Kuenemann, Olivier Sperandio, Laurent E. Dardenne, and Maria A. Miteva. 2021. New Machine Learning and Physics-Based Scoring Functions for Drug Discovery. Scientific Reports 11(1):3198. doi: 10.1038/s41598-021-82410-1.

Em 11 de março foram depositados em bases de dados públicos internacionais (GISAID) o resultado do sequenciamento de 195 genomas provenientes da colaboração de pesquisadores de cinco estados (AM, RN, PB, BA e RJ) representando 39 municípios brasileiros. Participaram dessa iniciativa o LNCC/MCTI, UFRN, UFPB, UESC e UERJ. Os resultados foram submetidos nessa mesma data para periódico científico internacional e foi assinado por 22 pesquisadores pertencentes a essas instituições.
O esforço foi organizado pelo Laboratório de Bioinformática (LABINFO) do Laboratório Nacional de Computação Científica (Petrópolis-RJ) um instituto do Ministério da Ciência e Tecnologia, Inovações (MCTI) com o objetivo de compreender a existência de variantes genéticas do SARS-CoV2 considerando que poucos genomas estão amostrados. Essa iniciativa faz parte da Rede Vírus, do MCTI/FINEP/CNPq, Rede Corona-ômica-RJ da FAPERJ e da CAPES.
As amostras coletadas foram de pessoas com resultados positivo para COVID-19 no período de 1 de dezembro de 2020 até 15 de fevereiro 2021. As pessoas tinham faixa etária heterogênea, variando de 11 a 90 anos de idade, sendo 92 homens e 93 mulheres.
Os resultados permitiram a inferência das datas de origem de P.1 (linhagem de Manaus) e identificar que a linhagem do Rio de Janeiro, P.2, descrita no final de 2020 apresenta diversificação à medida que se espalha pelo país. Além disso foi possível reconstruir as rotas de transmissão interestaduais dessas duas linhagens.
Importante ainda é salientar que com o sequenciamento de três amostras que contém a mutação E484K, o presente estudo permitiu a identificação de uma possível nova linhagem de SARS-CoV-2 originada da linhagem B.1.1.33 que circulava no Brasil desde o início de 2020. Esta contém agora a mutação E484K, na proteína S, associada ao escape imunológico e, portanto, pode ter implicações para planejamento de novas estratégias para o controle da COVID19.
Considerando que novas estratégias para controle da pandemia da COVID19 necessitam estudos colaborativos e em Rede, devido ao surgimento e a disseminação de novas variantes circulantes de SARS-CoV-2, fazemos este anúncio em grupo antes mesmo que o artigo seja revisado pelos pares.
Além disso, fica claro a necessidade crescente de uma vigilância genômica eficaz para poder precocemente identificar potenciais mutações virais auxiliando desta forma o aprimoramento das vacinas atuais contra COVID19.

Infraestrutura de computação de alto desempenho está disponível para suporte a atividades de ensino, pesquisa e desenvolvimento.
O supercomputador Santos Dumont do Laboratório Nacional de Computação Científica - LNCC (unidade de pesquisa vinculada ao Ministério da Ciência, Tecnologia e Inovações - MCTI), localizado em Petrópolis, Região Serrana do Rio de Janeiro, está com chamada aberta para submissão de novos projetos. O prazo para apresentação das propostas de uso é até o dia 27 de novembro. Além do SDumont, as propostas podem ser realizadas em conjunto com o supercomputador Lobo Carneiro - LoboC, instalado no Instituto de Pós-graduação e Pesquisa de Engenharia COPPE da Universidade Federal do Rio de Janeiro - UFRJ.
Os proponentes ao uso dos supercomputadores serão notificados sobre a aceitação do projeto à medida que as propostas forem avaliadas (em processo de fluxo contínuo). A previsão é de o processo de avaliação dure aproximadamente dois meses. Já a implementação tem prazo médio de 15 dias após a aprovação do projeto. Cada proposta enviada para avaliação deverá ser autocontida, justificando a necessidade de uso dos recursos e demonstrando clareza do objetivo almejado.
Os pesquisadores interessados em utilizar os recursos computacionais do SDumont e do LoboC devem (pesquisador principal) ter vínculo empregatício com instituição de ensino ou de pesquisa no Brasil, além de apresentar carta de anuência da instituição em que permanecerá, durante a vigência do projeto. São elegíveis profissionais pós-doutorandos, professor visitante, pesquisador visitante, pesquisador/professor especial (aposentado, mas que mantém algum vínculo não-remunerado com a instituição). Além disso, as propostas submetidas devem demonstrar relevância científica ou tecnológica, explicitando seus aspectos transformadores, o impacto científico esperado e as possíveis inovações e aplicações práticas.
O SDumont é uma ferramenta científica, que figura no ranking dos 500 supercomputadores mais rápidos do mundo (o mais rápido da América Latina com seus 5,1 petaflops, entre os supercomputadores voltados para o uso científico). Atualmente, cerca de 230 projetos de pesquisas, incluindo estudos sobre a exploração de petróleo e gás, carvão mineral, energias renováveis e fenômenos climáticos, além de pesquisas sobre o vírus Zika, HIV, Dengue e Coronavírus, utilizam a capacidade de processamento do supercomputador.
Tanto o SDmunont, quanto o LoboC fazem parte do Sistema Nacional de Processamento de alto Desempenho SINAPAD, uma infraestrutura de computação de alto desempenho disponível a instituições brasileiras, públicas ou privadas, para suporte a atividades de ensino, pesquisa e desenvolvimento. Todo pesquisador vinculado a uma instituição brasileira, com um problema relevante e que demande um sistema computacional de larga escala, pode submeter propostas para utilizar os recursos computacionais do SINAPAD. O SDumont é o nó principal dessa infraestrutura e, por esse motivo, apresenta um processo de avaliação de propostas próprio.
Para mais informações, acesse o link.

As inscrições devem ser feitas até o dia 9 de abril para o ingresso nos cursos de mestrado e doutorado stricto sensu.
Os interessados em realizar mestrado ou doutorado em Modelagem Computacional no Laboratório Nacional de Computação Científica - LNCC (unidade de pesquisa vinculada ao Ministério da Ciência, Tecnologia e Inovações - MCTI) podem se inscrever a partir do dia 1 de março. A Comissão de Avaliação e Seleção (CAS) do Programa de Pós-Graduação receberá as inscrições para o processo seletivo até o dia 9 de abril. Os aprovados terão de efetuar a matrícula nos períodos letivos que se iniciam em junho ou setembro.
A avaliação do curso de pós-graduação do LNCC junto à Área Interdisciplinar da Coordenação de Aperfeiçoamento de Pessoal de Nível Superior - CAPES (órgão do Governo Federal, vinculado ao Ministério da Educação - MEC) é o conceito seis, nota que aponta alto padrão internacional de desempenho, para programas de pós-graduação. A avaliação periódica dos cursos de pós-graduação (mestrados e doutorados) do país é realizada a cada quatro anos. O conceito máximo é sete.
O Programa de Pós-Graduação de Modelagem Computacional do LNCC foi iniciado em 2000 e congrega docentes de todas as Coordenações de Pesquisa e Desenvolvimento do LNCC, bem como colaboradores de outras instituições do País e do exterior. O Programa de Pós-Graduação em Modelagem Computacional do LNCC (PG-LNCC) visa prover uma formação multidisciplinar em Ciências Exatas, Ciências da Computação e Ciências da Vida a graduados em Matemática, Física, Química, Engenharias, Computação, Biologia, Economia ou outras áreas afins.
Para ingresso no Programa de Mestrado, o candidato deve ter concluído um curso de graduação em uma das referidas áreas. E para ingresso no Programa de Doutorado, o candidato deve ter concluído um curso de mestrado stricto sensu em uma das áreas citadas.
Para mais informações, acesse o site da pós graduação.

Fábio Borges de Oliveira
Wagner Léo
Sérgio Ferreira de Figueiredo - Coordenador
Matheus B. de Mendonça
Coordenação de Tecnologia da Informação e Comunicação - COTIC
Serviço de Suporte de Sistemas e Redes
Lígia Morais - Coordenadora do SECIN
Tathiana Tapajós, Graziele Soares - Equipe SECIN
Laboratório Nacional de Computação Científica - LNCC
Sala 2D10
Av. Getúlio Vargas, 333 - Quitandinha
Petrópolis - Rio de Janeiro - Brasil
CEP: 25651-075
Tel.: +55 (24) 2233 6249/6101
Email: secin@lncc.br
2594-8997

 PDF
PDF

