Boletins de notícias LNCC

O LNCC Notícias é um boletim digital online, de acesso público e periodicidade mensal, para divulgar as atividades e notícias do Laboratório Nacional de Computação Científica (LNCC/MCTIC). A reprodução parcial ou total das notas é autorizada, desde que acompanhada das devidas referências e créditos da publicação, indicando o link para a página.
Amostras foram coletadas no Sul do Brasil e depositadas em bases internacionais, além de submetidas, por meio de um estudo, em um periódico científico.
Uma semana depois da Organização Mundial da Saúde (OMS) demonstrar preocupação com as novas variantes da Covid-1 9, salientando a necessidade de fazer uma investigação meticulosa sobre possíveis impactos em vacinas, casos de reinfecções e coinfecções, pesquisadores apresentaram resultados de estudos de amostras coletadas no Rio Grande do Sul, onde foi possível a caracterização de cinco linhagens diferentes que estão circulando no estado, sendo uma nova, a princípio denominada VUI-NP13L, e dois casos de coinfecção. Os genomas sequenciados foram depositados em bases internacionais e o estudo submetido a um periódico científico.
O trabalho foi realizado pelo Laboratório de Bioinformática - Labinfo, do Laboratório Nacional de Computação Científica - LNCC, Petrópolis-RJ (unidade de pesquisa do Ministério da Ciência, Tecnologia e Inovações - MCTI), coordenado por Ana Tereza Vasconcelos, e pelo Laboratório de Microbiologia Molecular da Universidade Feevale (Novo Hamburgo-RS), coordenado pelo professor Fernando Spilki. A iniciativa faz parte da RedeVírus MCTI, da qual participam os dois laboratórios.
O material genético foi coletado pelo Laboratório de Microbiologia Molecular da Universidade Feevale, que recebe espécimes clínicos de pacientes de 40 municípios do Rio Grande do Sul. A amostragem, realizada por funcionários de saúde dessas cidades, foi enviada ao Laboratório juntamente com informações dos pacientes, como idade, sexo, sinais clínicos e possível cantata com outros casos suspeitos ou confirmados. Para esses estudos, foram selecionadas 92 amostras de pacientes com faixa etária de 14 a 80 anos de idade, sendo 50% homens e 50% mulheres.
Posteriormente, as amostras foram enviadas ao LNCC para a realização do sequenciamento genético e análises de bioinformática. Usando ferramentas desenvolvidas pelo grupo do Labinfo, foi possível a caracterização das cinco linhagens diferentes que estão circulando no Rio Grande do Sul. Entre elas, a nova linhagem, denominada pelos pesquisadores como VUI-NP13L. Também foram identificados dois casos de coinfecção, quando há a presença de duas linhagens de vírus em um mesmo indivíduo.
Possibilidade de dispersão do vírus
Os estudos realizados com amostras do Rio Grande do Sul geram preocupação devido à possibilidade de dispersão do vírus para outros Estados e países vizinhos da América do Sul. A Região Metropolitana de Porto Alegre concentra o maior número de casos. Por isso, a análise do genoma ajuda a entender melhor a dinâmica, a estrutura populacional e as cadeias de transmissão locais do vírus. Os pesquisadores também estão conduzindo experimentos in vitro na nova linhagem encontrada no estado, incluindo isolamento viral e investigação sobre neutralização ou anticorpos presentes no soro de pacientes infectados e recuperados.
Foi possível ainda confirmar a disseminação generalizada da variante E484K na proteína S, que é a mesma mutação do novo Coronavírus identificada no Rio de Janeiro no mês passado. "Isso é preocupante, pois sabe-se que essa mutação pode estar associada a um escape de anticorpos formados contra outras linhagens do vírus. É mais uma evidência que essas novas linhagens podem causar problemas mesmo em pessoas que já tenham uma imunidade prévia contra o Sars-Cov-2", afirma Fernando Spilki.
Outro ponto importante foi a identificação da coinfecção, que é uma infecção simultânea por vírus com genomas distintos em um mesmo indivíduo, e que foi encontrada em dois casos clínicos ocorridos no final de novembro. A coinfecção com a variante E484K não havia sido descrita até o momento.
A preocupação é porque a mistura de genomas de diferentes vírus coinfectando o mesmo indivíduo. A chamada recombinação, é um dos fenômenos que está na base da evolução de novo Coronavírus. "Mas, apesar da coinfecção, os dois pacientes tiveram um quadro de Covid-19 de leve a moderado e se recuperaram sem a necessidade de hospitalização", comentam os pesquisadores, lembrando a importância de se estabelecer medidas de distanciamento social para conter a propagação de novas variantes, potencialmente mais transmissíveis.
Segundo a coordenadora do Labinfo do LNCC, Ana Tereza Vasconcelos, os dados servem de alertar as autoridades sanitárias que estudos sobre a dispersão do Sars-Cov-2 são extremamente importantes para a segurança de todos.

Desde 2002 na instituição federal, o servidor de carreira assume um mandato de quatro anos.
O matemático Fábio Borges de Oliveira foi nomeado como novo diretor do Laboratório Nacional de Computação Científica - LNCC (unidade de pesquisa vinculada ao Ministério da Ciência, Tecnologia e Inovação - MCTI), para um mandato de quatro anos. A nomeação, publicada na forma de portaria, na quarta-feira (20), no Diário Oficial da União e assinada pelo ministro chefe da Casa Civil, Walter Souza Braga Netto, tem em vista o disposto no art. 4º do Decreto nº 9.794, de 14 de maio de 2019.
O Prof. Dr.-Ing. Fábio Borges assume o cargo que, desde 2015, vinha sendo ocupado pelo também servidor público federal Augusto César Gadelha, doutor em Engenharia Elétrica pela Universidade de Stanford, Estados Unidos, cuja trajetória com o LNCC foi iniciada em 1982. Durante 16 anos (até 1998) como pesquisador titular, exercendo ainda o cargo de diretor adjunto entre 1985 e 1997, além da chefia do departamento de pesquisa e desenvolvimento, nos períodos de 1985 a 1987 e 1989 a 1996.
Trajetória do novo diretor
Prof. Dr.-Ing. Fábio Borges de Oliveira tem trabalhado por muitos anos nas áreas de ciência e tecnologia. Em 1994, um concurso público selecionou-o para trabalhar na Universidade Estadual de Londrina (UEL). Em 2002, outro concurso público selecionou-o para trabalhar no MCTI, lotado no LNCC. Ele obteve o doutorado em Engenharia (Doktor-Ingenieur) pelo departamento de Ciência da Computação da Universidade Tecnológica de Darmstadt - Alemanha (TU Darmstadt - Technische Universität Darmstadt), o grau de mestrado em Modelagem Computacional no LNCC e o bacharelado em Matemática na Universidade Estadual de Londrina (UEL). Sua tese de doutorado tem o título On Privacy-Preserving Protocols for Smart Metering Systems, trata sobre protocolos de preservação de privacidade para sistemas de medição inteligentes em infraestruturas críticas. Na universidade alemã, foi ainda pesquisador assistente com bolsa da fundação Horst Görtz.
Atualmente, ele está desenvolvendo pesquisas em algoritmos, segurança, privacidade e redes neurais no LNCC e ministrando disciplinas como Curvas Elípticas e Hiperelípticas, Criptografia, Criptografia Homomórfica, Programação em Criptografia, Fundamentos da Ciência da Computação, Inteligência Artificial Aplicada à Segurança Cibernética, Números p-ádicos e Escrita Científica. Entre 2005 e 2007 foi ainda chefe do Setor de Treinamento e Apoio da Coordenação de Sistemas e Redes da instituição. Entre 2004 e 2009, professor do Instituto Superior de Tecnologia em Ciência da Computação de Petrópolis - ISTCC-P.
Entre os diversos trabalhos publicados, destaca-se o livro sobre Inteligência Artificial Statistical Learning Using Neural Networks: A Guide for Statisticians and Data Scientists with Python publicado pela Chapman and Hall/CRC em 2020 em coautoria com os professores Pereira, B. B. e Rao, C. R. Além disso, também o livro sobre segurança e privacidade On Privacy-Preserving Protocols for Smart Metering Systems: Security and Privacy in Smart Grids, publicado pela Springer International Publishing, em 2017.
Desde 2019, é coordenador do projeto de pesquisa Criptografia em Software de Modelagem Computacional Hidro-Geomecânica de Reservatórios desenvolvido no LNCC. Entre 2005 e 2009, coordenou a pesquisa Segurança da Informação, um estudo de algoritmos de criptografia e esteganografia para prover segurança a informações críticas. Desde 2002, é ainda integrante do Centro de Processamento de Alto Desempenho CENAPAD (vinculado ao Sistema Nacional de Processamento de Alto Desempenho SINAPAD), projeto do LNCC que tem como objetivo a utilização de serviços de computação de alto desempenho para atender ao meio acadêmico, empresarial e governamental no desenvolvimento de simulações tratamento de dados em áreas como meteorologia, petróleo, indústria automobilística e bioinformática.
Entre 2013 e 2019, foi revisor dos periódicos científicos como Security and Communication Networks, IEEE Transactions on Smart Grid, Journal of Cloud Computing: Advances, Systems and Applications, EURASIP Journal on Information Security, Future Generation Computer Systems, IEEE Communications Surveys and Tutorials, IEEE Transactions on Cloud Computing (TCC), Evolving Systems, Computer Journal (Print), IEEE Access, Applied Mathematics and Computation e Journal of Network and Computer Applications.
Premiações e reconhecimentos
2020 - Melhor Artigo do SBSeg - Criptografia, SBC.
2020 - Menção Honrosa do SBSeg - Artigos Curtos, SBC.
2019 - Melhor Artigo (WRAC+) SBSeg, SBC.
2018 - IEEE Senior Member, IEEE.
2018 - Latin America Distinguished Service Award, IEEE Communications Society.
2014 - Best Poster: Introducing asymmetric DC-Nets, IEEE - CNS.
2007 - 3º Melhor Artigo do WTICG do SBSeg, SBC.

Na chamada pública divulgada na última semana, foram aprovados 12 projetos com investimento total de R$ 7,2 milhões.
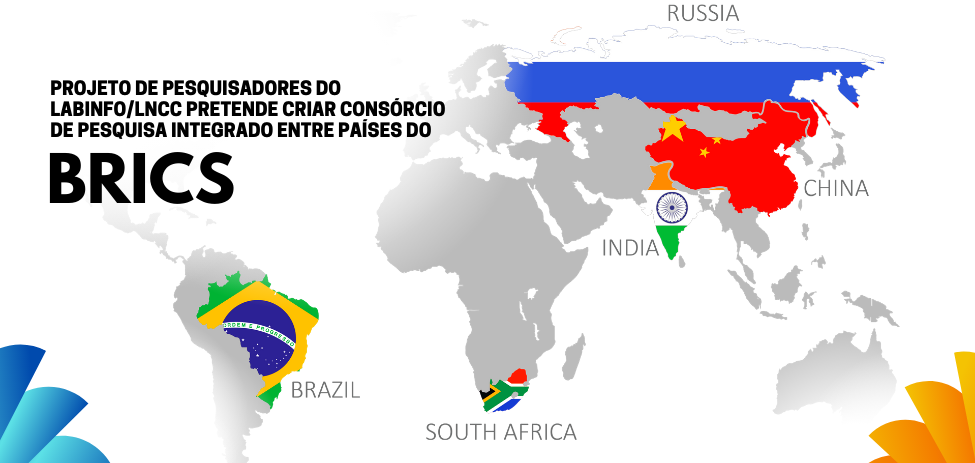
O projeto de combate à pandemia da Covid-19, da Rede para Vigilância Genômica de SARS-CoV-2, que inclui organizações líderes de pesquisa e saúde pública nos cinco países do BRICS (Brasil, Rússia, Índia, China e África do Sul - NGS-BRICS), foi aprovado, na última semana, na chamada pública da cooperação internacional. No Brasil, a rede será coordenada pelo Laboratório de Bioinformática - Labinfo, do Laboratório Nacional de Computação Científica - LNCC (unidade de pesquisa do Ministério da Ciência, Tecnologia e Inovações - MCTI). Integram ainda a proposta a Universidade Federal do Rio de Janeiro - UFRJ, a Universidade Feevale (Rio Grande do Sul), a Universidade Federal de Minas Gerais - UFMG e a Universidade Estadual Paulista - UNESP, membros da CoronaÔmica-BR, apoiada pelo MCTI. Na chamada pública realizada pelo Conselho Nacional de Desenvolvimento Científico e Tecnológico - CNPq, ao todo, 12 projetos foram aprovados para um investimento total de R$ 7,2 milhões.
"O objetivo geral deste projeto é o de criar um consórcio de pesquisa genômica integrado, interdisciplinar e colaborativo entre as principais organizações científicas do BRICS, para promover o conhecimento e monitoramento espacial e genômico da dispersão do vírus e contribuir para a melhoria do sistema de saúde dos países envolvidos", ressalta a coordenadora do Labinfo, do LNCC/MCTI, Ana Tereza Ribeiro de Vasconcelos.
Também são objetivos do projeto: aumentar o acesso aos métodos genômicos para sequenciar o SARS-CoV-2 nos países do BRICS, em tempo quase real, e traçar a evolução do vírus nesses países; rastrear as introduções do SARS-COV-2, identificar as transmissões na comunidade e usar essas informações para caracterizar e controlar surtos locais; usar, desenvolver e aplicar big data e métodos epidemiológicos para entender, controlar e prevenir surtos e melhorar o diagnóstico e a terapêutica; além de capacitar e treinar as equipes de pesquisadores do BRICS.
A coordenação geral da rede ficará sediada na África do Sul (UKZN), sob a coordenação do Dr. Túlio de Oliveira, bioinformata brasileiro, que trabalha há mais de 20 anos com surtos virais, incluindo HIV, Hepatite B e C, Chikungunya, Dengue, SARS-CoV-2, Zika e Vírus da Febre Amarela. Além do Brasil, a rede conta: na Rússia, com o Skolkovo Institute of Science and Technology (SIST) e o Vavilov Institute of General Genetics (VIGG); na China, inclui o Beijing Institute of Genomics (BIG), Chinese Academy of Sciences (CAS), China National Center for Bioinformation, National Institute of Biomedical Genomics (IPBG) e Institute of Pathogen Biology (IPB); na Índia, inclui o National Institute of Biomedical Genomics (NIBMG), Centre for BioSystems Science and Engineering (BSSE) e o Indian Institute of Science Centre for DNA Fingerprinting and Diagnostics (CDFD); na África do Sul, inclui o National Health Laboratory Services (NHLS), o National Institute of Communicable Diseases (NICD), as Universidades de KwaZulu-Natal (UKZN), Cape Town (UCT), Free State (UFS) e Stellenbosch (US).
No Brasil, são financiadores dos projetos aprovados na chamada pública divulgada pelo CNPq o MCTI, o Fundo Nacional de Saúde - FNS e o Fundo Nacional de Desenvolvimento Científico e Tecnológico - FNDCT. A chamada tem como objetivo apoiar projetos de Pesquisa, Desenvolvimento e Inovação - P,D&I, que contribuam significativamente para o desenvolvimento científico e tecnológico e a inovação do país, no âmbito da cooperação CNPq/MCTI/BRICS-STI, para fazer frente ao combate da pandemia do novo Coronavírus, nas seguintes áreas temáticas, como: pesquisa e desenvolvimento de novas tecnologias e ferramentas para diagnósticos da Covid-19; pesquisa e desenvolvimento de vacinas e medicamentos para Covid-19, incluindo o reposicionamento de medicamentos disponíveis; sequenciamento genético da SARS-CoV-2 e estudos sobre epidemiologia e modelagem; inteligência artificial, TICs e Computação de Alto Desempenho orientados à pesquisa para novos medicamentos, desenvolvimento de vacinas, tratamentos, testes clínicos e sistemas e infraestruturas de saúde relacionados à Covid-19; e estudos epidemiológicos e testes clínicos para avaliar a sobreposição SARS-CoV-2 e outras comorbidades, em especial Tuberculose.
O CNPq recebeu 87 propostas consideradas qualificadas para serem avaliadas pelo Comitê de Avaliação Nacional, formado para esse propósito. Essas propostas foram avaliadas posteriormente pelo Comitê Internacional das Agências de Fomento do BRICS, sendo selecionadas propostas de consórcios formados por, pelo menos, três países do BRICS. O Brasil foi o único país do grupamento BRICS a ter participante em todos os consórcios aprovados na Reunião do Comitê Internacional das Agências do BRICS. Foram contemplados projetos nas grandes áreas do conhecimento de Ciências Biológicas, Ciências da Saúde, Ciências Exatas e da Terra, e Engenharias; nos estados de Goiás, Paraíba, Pernambuco, Rio de Janeiro e São Paulo.
Amostras coletadas no início de dezembro indicam a presença da variante E484K, mesma mutação do coronavírus identificada no Rio de Janeiro no mês passado e mais uma variante inédita no Rio Grande do Sul.
Na última sexta-feira (8), pesquisadores do Projeto Corona-ômica.Br MCTI, integrantes da RedeVírus MCTI, emitiram um comunicado em que alertam autoridades sanitárias sobre a descoberta, em análise preliminar, da circulação da nova linhagem do novo coronavírus originária do RJ também no Rio Grande do Sul. O projeto tem participação do Laboratório Nacional de Computação Científica (LNCC), unidade de pesquisa vinculada ao Ministério da Ciência, Tecnologia e Inovações (MCTI), juntamente com o Laboratório de Microbiologia Molecular da Universidade Feevale de Novo Hamburgo (RS).
Os pesquisadores, que realizam a análise genômica de 92 amostras virais de circulação recente no estado, identificaram a presença, na proteína S, da variante E484K, a mesma encontrada em mutação em algumas cidades do Rio de Janeiro. Além dessa, foi identificada uma nova linhagem inédita com outras mutações circulando no RS. A nota assinada por Fernando Rosado Spilki, coordenador da Rede Corona-ômica.BR MCTI, da Universidade Feevale e Ana Tereza Vasconcelos, Coordenadora do Laboratório de Bioinformática - LNCC/MCTI, afirma que os genomas sequenciados foram depositados em bases internacionais e serão descritos em publicação científica.
"Com o objetivo de colaborar com a comunidade científica no enfrentamento da Covid-19, os genomas sequenciados foram depositados em bases internacionais e em breve a caracterização destas amostras serão descritas em publicação científica de amplo acesso, diz o texto.
Os cientistas ainda destacaram a importância de redes colaborativas para monitorar as variações do vírus e traçar estratégias. A crescente diversidade genética do SARS-CoV-2 encontrada nesse estudo e em trabalhos recentes no Brasil reitera a importância da constituição de redes de sequenciamento e análises genéticas colaborativas para a realização de vigilância genômica, da necessidade de investimento em pesquisa e formação de recursos humanos de forma contínua para que possamos enfrentar esta pandemia e futuros desafios de saúde pública no país".
Ambas unidades de pesquisa envolvidas no novo estudo estão ligadas às Rede Corona-ômica.BR MCTI (Rede Nacional de ômicas de Covid-19 para identificação de fatores associados à dispersão da epidemia e severidade), uma iniciativa da RedeVírus MCTI, um comitê de especialistas criado em fevereiro de 2020 por portaria do MCTI para traçar estratégias de combate ao vírus com o uso da estrutura científica do país.

O material, disponível gratuitamente para a comunidade científica, servirá para estudar a expansão acelerada do universo e o fenômeno da energia escura. Os dados serão processados no Supercomputador Santos Dumont, do LNCC.
O Dark Energy Survey (DES), em colaboração com o Fermilab (EUA), o National Center for Supercomputing Applications (NCSA, EUA), o NOIRLab (EUA) e com o Laboratório Interinstitucional de e-Astronomia LIneA (Brasil), vai tornar público hoje (14), através de um evento online, para toda a comunidade científica, seu catálogo de imagens e dados astronômicos coletados ao longo dos últimos seis anos. Esta é a segunda liberação pública de dados do DES (DR2), uma coletânea de imagens e objetos identificados com o objetivo de compreender a expansão acelerada do universo e o fenômeno da energia escura, cobrindo 5.000 graus quadrados no Hemisfério Sul (um oitavo de todo o céu).
As novidades envolvendo o DR2 são quase 700 milhões de objetos astronômicos - com base nos 400 milhões já catalogados com o lançamento anterior (DR1) e o refinamento da técnica de calibragem, o que melhorou a qualidade e a estimativa da distribuição de matéria no Universo. Os dados coletados serão disponibilizados em milhares de imagens do céu, e também no formato de catálogos dos objetos referentes a estas imagens.
Além destas pesquisas que, complementam os resultados científicos já alcançados pela colaboração, o DR2 vai permitir muitas outras pesquisas. Um exemplo é a possível descoberta de novos objetos do Sistema Solar e a investigação da natureza das primeiras galáxias, explicando como as primeiras estrelas do Universo foram formadas, além de vínculos importantes sobre a matéria escura. O DR2 é um dos maiores catálogos astronômicos liberados publicamente até hoje.
O DR2 é o resultado de um grande esforço do LineA, na forma de uma plataforma onde o usuário pode acessar as imagens coadicionadas do levantamento do DES, os catálogos de objetos e mapas, além da possibilidade de serem efetuadas buscas nos catálogos principais do DR1 e DR2. O site, que já estava disponível para a primeira liberação de dados do DES, foi completamente reformulado, de forma a facilitar o trabalho do cientista. Além das funções triviais de acesso e visualização das imagens e buscas no banco de dados, o usuário, por exemplo, pode inserir uma lista de posições de objetos, inspecioná-los, e depois baixar estas imagens. Outra função é a possibilidade de comparar campos específicos nas imagens das duas versões (DR1 e DR2) ou selecionar objetos, via busca no banco de dados, e diretamente inspecionar cada objeto - comparando com outros levantamentos públicos (2MASS, SDSS, Gaia EDR3, etc), além de buscar informações sobre objetos próximos no banco de dados do NASA Extragalactic Database (NED) ou no VizieR.
O trabalho foi completamente desenvolvido pela equipe do LIneA, onde o time de TI interagiu com os cientistas na coordenação do projeto. Contou ainda com a interação das equipes do NCSA e a colaboração do DES na busca por soluções práticas para os cientistas. Além deste serviço desenvolvido pelo LIneA e instalado nas máquinas do NCSA em Illinois, EUA, a instituição brasileira trabalha para que, nos próximos meses, seja instalada uma versão pública hospedada no Brasil, com acesso às imagens e catálogos. Um grande esforço, desde a transferência dos dados no NCSA até a instalação do servidor e disponibilização das máquinas para o uso público. Vale ressaltar que o volume de dados é muito grande e seria inviável para pesquisadores individuais ou organizações terem acesso ao conjunto de dados sem as estruturas e ferramentas necessárias.
"A disponibilização pública dos dados de seis anos do Dark Energy Survey, conhecido como DR2, aqui pelo LIneA, é mais um importante passo na implantação de um Centro de Dados Astronômicos no Brasil. Já fazemos isso para outro importante levantamento - o Sloan Digital Sky Survey há mais de 10 anos - e estamos pleiteando hospedar os dados do Legacy Survey of Space and Time, que fará um filme da metade do céu durante 10 anos registrando variações de brilho e posição de objetos, além de um censo dos objetos do sistema solar. Um dos maiores benefícios de ter esses acervos no LIneA é ter os dados próximos da infraestrutura de processamento do LIneA e do supercomputador Santos Dumont para facilitar a análise científica por pesquisadores brasileiros evitando a transferência de grandes volumes de dados através de conexões internacionais ou depender de acesso a centros de computação em outros países." disse Luiz Nicolaci da Costa, diretor do LIneA.
Os dados completos do DR2 estão online e disponíveis para que o público faça suas descobertas no site disponibilizado pelas instituições na cooperação internacional. O LIneA agradeceu ainda o apoio do Laboratório Nacional de Computação Científica - LNCC, Petrópolis, Rio de Janeiro (unidade de pesquisa vinculada ao Ministério da Ciência, Tecnologia e Inovações MCTI), onde está instalado o cluster de computadores do LIneA, da Rede Nacional de Ensino e Pesquisa pelo suporte na transmissão dos dados e do Instituto Nacional de Ciência e Tecnologia (INCT) do e-Universo pelo suporte com pessoal e equipamento.
O LIneA e o INCT do e-Universo tem como missão apoiar a participação de pesquisadores associados a instituições brasileiras em grandes levantamentos astronômicos como o Dark Energy Survey (DES), Sloan Digital Sky Survey (SDSS), Dark Energy Spectroscopic Instrument (DESI), e o Legacy Survey of Space and Time (LSST). O LIneA é um instituto de ciência e tecnologia privado cuja missão é viabilizar a participação de pesquisadores e estudantes em colaborações internacionais; apoiar centros emergentes, fornecer acesso a acervos de dados astronômicos e a uma infraestrutura de processamento intensivo de dados, e desenvolver soluções para problemas de big data nas áreas de astronomia e cosmologia. Atualmente as atividades do LIneA são apoiadas pela FINEP, e CNPq e FAPERJ através do programa dos INCTs.

Os empreendimentos selecionados poderão se instalar no LNCC e contar com apoio da incubadora para o desenvolvimento do seu negócio.
A Administração da Incubadora do Laboratório Nacional de Computação Científica - LNCC/MCTI (unidade de pesquisa vinculada ao Ministério da Ciência, Tecnologia e Inovações) divulgou a primeira chamada pública de 2021 para a inscrição de novos empreendimentos inovadores e projetos de base tecnológica. Para participar do processo é preciso demonstrar potencial inovador, viabilidade técnica e mercadológica e ter afinidade com a pesquisa desenvolvida no LNCC.
"Nosso edital é de fluxo contínuo. Estamos fazendo esta chamada para avaliar as propostas recebidas, mas caso não sejam aprovadas ou ainda assim restem vagas para novas empresas, reabriremos o edital ao término desta seleção. Nossa missão é tirar projetos do papel e transformar conhecimentos em novos produtos, serviços e processos. Incentivando a inovação tecnológica e a transferência de tecnologia estimulando assim o sucesso de projetos inovadores no mercado nacional e internacional", destaca a secretária da Incubadora do LNCC, Ana Cláudia Perigolo Moura.
A Incubadora LNCC estimula o fomento da cultura empreendedora e da inovação regional em áreas compatíveis com a atuação de ensino, pesquisa e extensão do Instituto de Pesquisas do MCTI. O procedimento para inscrição, que encerrará no dia 20 de janeiro, pode ser conferido no site. A avaliação dos projetos será remota por causa das restrições impostas pela Covid-19 e deverá ocorrer no primeiro bimestre de 2021.
Os empreendimentos selecionados poderão usufruir dos serviços oferecidos pela Incubadora LNCC, como capacitação, assessoria técnico-administrativa para desenvolvimento e aperfeiçoamento dos processos de gestão de negócios, apoio técnico e tecnológico para desenvolvimento de produtos e processos inovadores, além da infraestrutura física e tecnológica.

Desde janeiro, os cursos estão sendo oferecidos de forma online para o público em geral.
O Laboratório Nacional de Computação Científica - LNCC/MCTI (unidade pesquisa vinculada ao Ministério da Ciência, Tecnologia e Inovações), localizado em Petrópolis, na Região Serrana do Rio de Janeiro, realiza, desde o dia 11 de janeiro, mais uma edição do Programa de Verão, a 19ª. Até o dia 11 deste mês, estão sendo oferecidos minicursos em temas como Modelagem Computacional, Computação Quânticas e Integrações de Redes Neurais.
Na última edição do evento, ainda presencial, foram mais de 300 participantes. Em 2021, em função da pandemia da Covid-19, a edição se tornou virtual, ocorre através de vídeo aulas na internet. Nada que tenha reduzido o interesse do público. Foram mais de 1.300 inscrições efetuadas. Número quatro vezes maior do que no ano passado. A inscrição foi realizada de forma gratuita.
Mais detalhes sobre a programação completa do evento estão disponíveis na nova página do PV2021 http://www.verao2021lncc.kinghost.net. O material está disponível também no Youtube.
Programação
- Semana Supercomputador SDumont - (11 a 22 de janeiro de 2021)
- VII Encontro em Modelagem Matemática do Crescimento Tumoral (25 a 29 de janeiro de 2021)
- Jornada em Ciência de Dados (01 a 05 de fevereiro de 2021)
- XIV Encontro Acadêmico de Modelagem Computacional - (08 a 10 de fevereiro de 2021)
- Jornada de Iniciação Científica e Tecnológica - (11 de fevereiro de 2021)
Minicursos
- Introdução à Computação Quântica - (25 a 28 de janeiro de 2021)
- Difusões Estocásticas com Comutação: Aplicações, Métodos Numéricos e
- Controle Ótimo (28 de janeiro de 2021)
- Fundamentos da Teoria de Aprendizado de Máquina (28 a 29 de janeiro de 2021)
- Organização e Informação (Biológicas) (01 a 05 de fevereiro de 2021)
- Integrando Redes Neurais, Modelos Físicos e Rendering para Animação de Fluidos (02 a 05 de fevereiro de 2021)

Segundo análise, a nova linhagem foi identificada em 38 dos 180 genomas sequenciados.
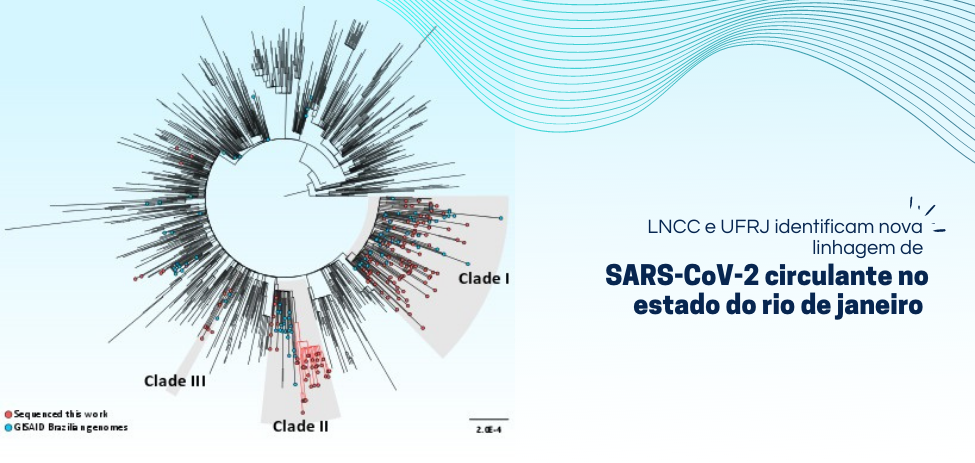
O Laboratório de Bioinformática - Labinfo, do Laboratório Nacional de Computação Científica - LNCC/MCTI (unidade de pesquisa do Ministério da Ciência, Tecnologia e Inovações), identificou cinco mutações, caracterizando uma possível nova linhagem originária do B.1.1.28 do novo Coronavírus. A análise, conduzida em colaboração com o Laboratório de Virologia Molecular da Universidade Federal do Rio de Janeiro - UFRJ, as Secretarias de Saúde de Maricá e do Rio de Janeiro e o Laboratório Central de Saúde Pública Noel Nutels sequenciou 180 genomas do SARS-CoV-2, provenientes de amostras do Estado do Rio de Janeiro.
A B.1.1.28 já circulava no Brasil no início do ano. As mutações são no C100U, C28253U, G28628U, G28975U e C29754U. Além dessas cinco, a mutação G23012A (E484K), no domínio de ligação ao receptor da proteína Spike, está amplamente espalhado nos genomas dessa linhagem. E484K foi anteriormente associada ao escape de anticorpos neutralizantes contra SARS-CoV-2.
A pesquisadora Ana Tereza Ribeiro de Vasconcelos, que é geneticista e bioinformata com doutorado em Genética pela UFRJ e que tem experiência na área de Bioinformática e de Biologia Computacional, atuando principalmente na integração de dados das várias camadas multi-ômicas através da computação de alto desempenho, além de responsável pelo Labinfo do LNCC, explica que de acordo com as análises filogenéticas, o surgimento desta nova linhagem ocorreu em julho de 2020 e foi identificada, principalmente, no Rio de Janeiro, em Cabo Frio, Niterói e Duque de Caxias na Baixada Fluminense.
Segundo Ana Tereza não existe indicação que essa linhagem seja mais transmissível ou que possa interferir na efetividade das vacinas que estão sendo desenvolvidas. Entretanto, ela ressalta a importância de estudos contínuos de vigilância genômica para análise da dispersão dessa nova linhagem e na identificação de novas variantes do SARS-CoV-2 no estado do Rio de Janeiro e no Brasil.
As análises indicam que a linhagem B.1.1.28 aparece como emergente, sendo identificada em 38 dos 180 genomas sequenciados. Por outro lado, os pesquisadores apontam que a linhagem B.1.1.33 está em declínio.
O trabalho foi financiado pela Faperj, Ministério da Ciência, Tecnologia e Inovações submetido em 20 de dezembro ao MedRxiv. Confira o artigo publicado: https://www.medrxiv.org/content/10.1101/2020.12.23.20248598v1
Fábio Borges de Oliveira
Wagner Léo
Sérgio Ferreira de Figueiredo - Coordenador
Matheus B. de Mendonça
Coordenação de Tecnologia da Informação e Comunicação - COTIC
Serviço de Suporte de Sistemas e Redes
Lígia Morais - Coordenadora do SECIN
Tathiana Tapajós, Graziele Soares - Equipe SECIN
Laboratório Nacional de Computação Científica - LNCC
Sala 2D10
Av. Getúlio Vargas, 333 - Quitandinha
Petrópolis - Rio de Janeiro - Brasil
CEP: 25651-075
Tel.: +55 (24) 2233 6249/6101
Email: secin@lncc.br
2594-8997

 PDF
PDF

